DNA代码中实现的3D引擎
使用移液器对每个试管的位置(行和列)进行编码以开始计算。
q = 0.01cxtm = 0.606axp = 0.606cytm = 0.898ayp = 0.898cztm = 1.243azp = 1.243mxyzm = 0.3nx = 0.036 + 0.555 Col + 0.147 Rowny = 0.853 + -0.517 Rownz = 0.737 + -0.270 Col + 0.302行
选择您喜欢的颜色的荧光团,并将其连接到链R0,以便在产生R物种时激活。
使用特定波长的光源(取决于您选择的荧光团)来渲染结果。
脚趾介导的链置换技术的简化动画(基于[2]的补充材料)
单个反应所需的寡核苷酸类型(基于[2]的补充材料)
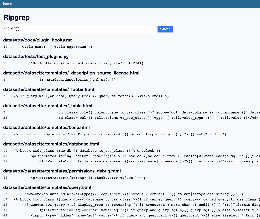
此实现的最小化源代码,仅包含带有几个宏的简单反应。缩小器是用Wolfram语言编写的,专门用于该项目。
最小化后,此项目中使用了10个反应。由于Piperine编译器在右侧不支持2种以上的产品,因此反应不一。
David Soloveichik,Georg Seelig和Erik Winfree DNA作为化学动力学的通用底物,《美国国家科学院院刊》,2010年3月,107(12)5393-5398; DOI:10.1073 / pnas.0909380107
Niranjan Srinivas,James Parkin,Georg Seelig,Erik Winfree和David Soloveichik无酶核酸动力学系统Science 358,eaal2052(2017)。一些图像是从补充材料中拍摄的
粉笔,卡梅伦,尼尔斯·科纳鲁普,怀亚特·里夫斯和戴维·索洛维奇克。连续化学反应网络中可组合的速率无关计算。系统生物学计算方法国际会议,第256-273页。占卜·斯普林格,2018年。
Chen,Ho-Lin,David Doty和David Soloveichik。连续化学反应网络中与速率无关的计算。第五届理论计算机科学创新会议论文集。 2014。
Marko Vasic,David Soloveichik和Sarfraz Khurshid CRN ++:分子编程语言自然计算(2020)19:391–407 DOI:10.1007 / s11047-019-09775-1 GitHub上的CRN ++